Home | Kontakt | Stellen | Publikationen | Links
Struktur und Funktion von Proprotein-Convertasen
Zahlreiche sekretorische und Membran-gebundene Proteine werden in der Zelle als Vorläufer-Proteine hergestellt (Abb. 1a). Diese s.g. Pro-Proteine müssen zunächst aktiviert werden, um eine biologische Aktivität zu entfalten. Dieser Vorgang wird oft durch limitierte Proteolyse reguliert, wobei ein Teil des Zielproteins hochspezifisch durch eine Protease abgespalten wird. Alternativ können Enzyme durch limitierte Proteolyse aber auch inaktiviert werden. Limitierte Proteolyse gehört neben Glycosylierungen, Phosphorylierungen und anderen chemischen Modifikationen zu den post-translation Modifikationen, die Proteine durchlaufen können. Die Proprotein-Convertase, eine Gruppe von Serin-Proteasen, spielen eine herausragende Rolle bei der proteolytischen Aktivierung einer Vielzahl von Pro-Proteinen.
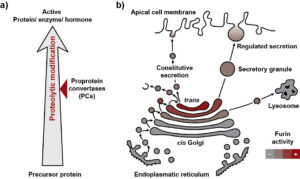
Abbildung 1 Physiologische Funktion der Proprotein-Convertasen
Säugetiere besitzen 9 verschiedene Vertreter der PCs, die sich durch stringente Sequenzspezifität auszeichnen. Dabei erkennen diese Proteasen typischerweise mehrere Aminosäuren innerhalb des Erkennungsmotivs ihrer Substrate. Furin kann als prototypischer Vertreter einer Untergruppe der PCs angesehen werden, die Substrate C-terminal poly-basischer Erkennungssequenzen des Typs R-X-(K/R)-R↓ (X steht für eine beliebige Aminosäure, K/R steht für Lysin oder Arginin an dieser Position, „↓“ symbolisiert die Peptidbindung, nach der gespalten wird) spalten. Durch die hohe Sequenzspezifität der PCs wird sichergestellt, dass ausschließlich die Zielproteine gespalten werden und eine unkontrollierte proteolytische Degradation der Proteine des sekretorischen Apparats verhindert wird.
Darüber hinaus gilt es für die Zelle zu verhindern, dass Enzyme, Rezeptoren oder Wachstumsfaktoren am falschen Ort oder zur falschen Zeit aktiviert werden. Daher muss gibt es verschiedene Mechanismen, um die Aktivität der PCs zu regulieren. Einerseits werden PCs in bestimmte Kompartimente des sekretorischen Apparats transportiert. Andererseits werden die PCs selbst als inaktive Proproteine synthetisiert und nach Erreichen des Zielkompartiments auto-aktiviert. Dabei erfolgt eine Serie auto-proteolytischer Spaltungen in der Prodomäne. Diese Prodomäne, dient dabei als Sensor, der die Autoaktivierung auslöst, sobald im Zielkompartiment ein mehr oder weniger saurer pH-Wert vorliegt. So wird Furin bei pH 6.0 des späten Golgi aktiviert (Abbildung 1b). Durch Transportprozesse gelangt Furin auch in sekretorische Vesikel und an die Zelloberfläche, von wo es wieder recycelt werden kann.
Grundlagen der Aktivierung von Proprotein-Convertasen
Im Rahmen eines vom Österreichischen Wissenschaftsfonds (FWF) geförderten Projekts (P 36648) untersuchen wir die Mechanismen der Auto-Aktivierung von Proprotein-Convertasen (PCs). Hierbei interessieren uns besonders strukturelle Änderungen dieser Proteasen, die während des Aktivierungsprozesses auftreten. Zudem unterscheiden sich die PCs in ihrem Aktivierungsprofil. Im Rahmen des Projekts sollen die Unterschiede der Aktivierungsmechanismen verschiedener PCs näher untersucht werden. Stellenausschreibungen zum Projekt finden Sie hier.
Mechanismen der Substraterkennung und Katalyse
PCs besitzen im Vergleich zu anderen Proteasen ein besonders hohe Substratspezifität. Hierbei erkennen die Proteasen mehrere positiv geladene Aminosäuren im Erkennungsmotiv ihrer Substrate. Proteinen und Peptide mit einzelnen Argininen oder Lysinen werden jedoch nicht von PCs gespalten. Wir interessieren uns für die Wechselwirkungen der Aminosäuren des poly-basischen Erkennungsmotivs mit der Substratbindetasche als Grundlage dieser Selektivität (Abb. 2). Detaillierte Kenntnisse dieser Mechanismen sind auch wertvoll für die Struktur-basierte Entwicklung besserer Inhibitoren (siehe unten).
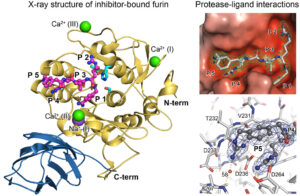
Abbildung 2 Kristallstruktur von Inhibitor-gebundenen Furin und Protease-Inhibitor-Interaktionen.
Strukturelle Untersuchungen haben gezeigt, dass die Substratbindetasche von Furin eine hohe Plastizität besitzt und in mehreren spezifischen Konformationen vorliegen kann. Wir konnten z.B. zeigen, dass es durch Bindung von Liganden zu einer Konformationsänderung der Substratbindetasche vom s.g. „OFF-state“ zum „ON-state“ kommt. Diese Konformationsänderung wirkt sich aber auch auf das katalytische Zentrum und andere strukturelle Bereiche von Furin aus.
Eine weitere spezifische Strukturänderung in der Substratbindetasche von Furin wird z.B. durch die Bindung Dichlorophenylpyridin-basierter Inhibitoren ausgelöst (Abb. 3). Der chlorierte Phenyl-Rest verdrängt hierbei eine Tryptophan-Seitenkette und bindet in eine s.g. kryptische Bindetasche. Interessanterweise zeigen sich fernab der Substratbindetasche ähnliche strukturelle Eigenschaften, die auch beim Übergang vom „OFF-state“ zum „ON-state“ zu finden sind.
Wir interessieren besonders für diese Konformationsdynamik von Furin und die molekularen Schaltmechanismen, die die katalytische Aktivität von Furin regulieren.

Abbildung 3 Kristallstruktur von Furin in Komplex mit einem Dichlorophenylpyridin-basierten Inhibitor.
Viele Erkrankungen, ein Wirkstoff – Entwicklung von Furin-Inhibitoren
Furin ist ein vielversprechendes Zielmolekül für die Entwicklung von Wirkstoffen gegen verschiedene Krankheiten. Wir entwickeln in Kooperation mit Chemikern und Virologen Inhibitoren, die z.B. für die Behandlung von Infektionskrankheiten Anwendung finden können.




